被子植物有超过30万个物种,最典型的特征之一是拥有形态各异的花。花可能与授粉昆虫协同演化,在被子植物的多样化过程中起重要作用,花重要形态特征如对称性的演化历史和相关基因的分子进化是进化生物学的研究热点之一。菊科是被子植物最大的两个科之一,拥有16个亚科、45个族及约190个亚族和亚族水平分支,包含大约28,000个物种,在全世界各地广泛分布,是多种生态系统的重要组成部分。许多菊科植物有重要的经济价值,如油料作物向日葵和广泛栽培的生菜;菊科有大量的观赏植物,如菊花、非洲菊、雏菊等;也有一些重要药用植物,如黄花蒿、菊花、红花、白术、洋甘菊等。菊科植物拥有由小花组成的头状花序,有的类型的小花是辐射对称的,而另一些类型则是两侧对称的。有超过15,000种菊科植物拥有两侧称的小花,其花冠伸长用以吸引传粉者,可能在菊科植物的多样化过程中起到了重要作用。因此,研究菊科花对称性的演化及其分子基础对认识菊科植物以及被子植物的多样化有重要意义。这样的进化生物学研究需要广泛取样的高分辨率系统发育树作为框架。之前的菊科核基因系统发育组学研究取样约250个物种解决了大部分亚科和族之间的关系,然而一些亚族之间的系统发育关系没有解决,还有很多亚族没有取样,限制了我们对菊科进化历史的认识,也影响了对菊科花对称性和其他重要性状演化模式及分子基础的研究。
近日,我院植物学系青年教师张国进博士及其合作者在植物学知名期刊《Plant Communications》(IF2022=10.5)以湖南师大为第一单位发表了题为“Nuclear phylogenomics of Asteraceae with increased sampling provides new insights into convergent morphological and molecular evolution”(doi: 10.1016/j.xplc.2024.100851)的研究论文。该研究基于300个样本模拟数据集和706个菊科物种真实数据集的分析,探索了一种整合部分转录组和低覆盖度浅层测序基因组数据获取单低拷贝核基因进行系统发育基因组学研究的新策略。结果显示在浅层测序基因组数据之外,增加占总物种数30%~50%的转录组数据,可以获得足够数量的单低拷贝核基因,基于这些基因重建的系统发育树分辨率高,与全部使用转录组数据获得的系统树在分辨率和拓扑结构方面差别很小。菊科真实数据集覆盖了144个亚族等级分支,其中43%的物种拥有转录组,其余物种仅拥有覆盖度为0.13X至1.5X的浅层测序基因组。基于整合的数据集获得了1,094个单低拷贝核基因序列并重建了菊科系统发育关系,很好地解决了绝大多数亚科、族和亚族之间的关系,许多亚族内关系也得到了高度支持,并发现了一个新的亚科水平分支和一个新的族水平分支。
此外,该研究基于新获得的菊科系统发育树探究了头状花序排列方式、花色、花对称性和冠毛类型等四个重要繁殖性状在菊科的演化历史,结果显示四个性状都在菊科内发生过多次平行演化。其中两侧对称花在菊科平行地起源了7次(图1),同时也发现了多次花两侧对称性丢失事件。对MADS-box家族参与ABCE模型基因的进化研究显示,这些基因在菊科祖先和科内多次重复,可能与多个繁殖性状的平行演化相关。分子进化和表达分析显示调控花对称性的CYC2基因在菊科发生过多次重复,CYC2基因的多个拷贝在菊科不同起源的两侧对称花类群中表达模式发生了分化。另外,多个CYC2基因低表达或不表达与部分分支花两侧对称性的丢失可能有密切联系。这些结果显示CYC2基因不同拷贝表达模式的分化可能是菊科两侧对称花平行演化的重要分子基础。
总之,该研究展示了一种整合物种转录组数据和低覆盖度浅层测序基因组数据进行大规模系统发育基因组学研究的新策略,构建了高分辨率菊科核基因系统发育树,提出了菊科重要繁殖性状特别是花两侧对称性的平行演化模式和分子基础的新见解。
(原文链接:https://www.cell.com/plant-communications/fulltext/S2590-3462(24)00080-4)
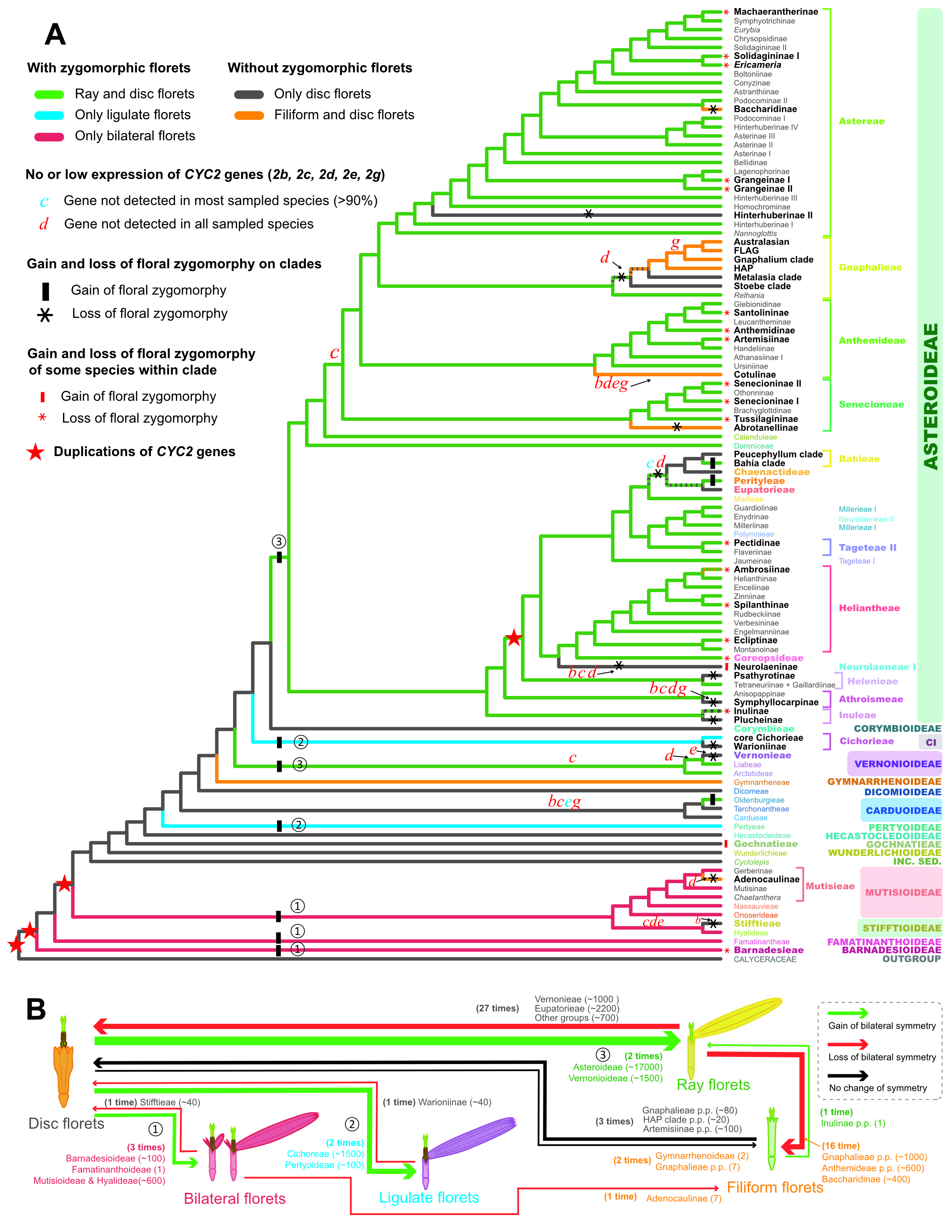
图1 (A)基于转录组和超低覆盖度浅层测序基因组数据获取的1094个核基因重建的菊科核基因系统发育树,展示花对称性在菊科的平行演化模式和CYC2基因的低表达或不表达与花两侧对称性丢失的联系。(B)通过祖先性状重建获得的菊科不同类型小花之间的演化模式。


